问题描述
我有 18 对变量,我想对它们进行成对数学运算以计算 18 个新变量。将公式应用于一列时,dplyr 中的 cross() 函数非常方便。有没有办法将 cross() 应用于成对的列?
简单除法 2 个变量的小例子(我的实际代码会更复杂,一些 ifelse,...):
library(tidyverse)
library(glue)
# filler data
df <- data.frame("label" = c('a','b','c','d'),"A" = c(4,3,8,9),"B" = c(10,4,1),"error_A" = c(0.4,0.3,0.2,0.1),"error_B" = c(0.3,0.4,0.1))
# what I want to have in the end
# instead of just 2 (A,B),I have 18
df1 <- df %>% mutate(
'R_A' = A/error_A,'R_B' = B/error_B
)
# what I'm thinking about doing to use both variables A and error_A to calculate the new column
df2 <- df %>% mutate(
across(c('A','B'),~.x/{HOW DO I USE THE COLUMN WHOSE NAME IS glue('error_',.x)}
.names = 'R_{.col}'
)
解决方法
一个选项是map/reduce。指定感兴趣的列 ('nm1'),在 map 中循环它们,select 来自数据集的那些列,reduce 通过划分,rename 列绑定后的列(_dfc),并将它们与原始数据集绑定
library(dplyr)
library(purrr)
library(stringr)
nm1 <- c('A','B')
map_dfc(nm1,~ df %>%
select(ends_with(.x)) %>%
reduce(.,`/`) ) %>%
rename_all(~ str_c('R_',nm1)) %>%
bind_cols(df,.)
-输出
# label A B error_A error_B R_A R_B
#1 a 4 10 0.4 0.3 10 33.33333
#2 b 3 0 0.3 0.0 10 NaN
#3 c 8 4 0.2 0.4 40 10.00000
#4 d 9 1 0.1 0.1 90 10.00000
或者带有 across
df %>%
mutate(across(c(A,B),~
./get(str_c('error_',cur_column() )),.names = 'R_{.col}' ))
# label A B error_A error_B R_A R_B
#1 a 4 10 0.4 0.3 10 33.33333
#2 b 3 0 0.3 0.0 10 NaN
#3 c 8 4 0.2 0.4 40 10.00000
#4 d 9 1 0.1 0.1 90 10.00000
我喜欢上面的 akruns 答案,尤其是带有 cur_column() 的方法。有趣的是,cur_column() 不能与 {rlang} 的求值 (!! sym(paste0("error_",cur_column()))) 一起使用,但 get 是一个不错的解决方法。
再添加一种方法,它也适用于 dpylr mutate 自定义函数与 purrr::reduce() 一起使用。在此函数中,x 是您的字符串词干,您可以使用 !! sym(paste0(...)) 构造要访问的所有变量。在左侧,您可以使用 {rlang} 的粘合语法。
您通过对字符串向量调用 reduce() 来应用此自定义函数,并且您的 data.frame 进入 .init = . 参数。
library(tidyverse)
library(glue)
# filler data
df <- data.frame("label" = c('a','b','c','d'),"A" = c(4,3,8,9),"B" = c(10,4,1),"error_A" = c(0.4,0.3,0.2,0.1),"error_B" = c(0.3,0.4,0.1))
gen_vars1 <- function(df,x) {
mutate(df,"R_{x}" := !! sym(x) / !! sym(paste0("error_",x)))
}
df %>%
reduce(c("A","B"),gen_vars1,.init = .)
#> label A B error_A error_B R_A R_B
#> 1 a 4 10 0.4 0.3 10 33.33333
#> 2 b 3 0 0.3 0.0 10 NaN
#> 3 c 8 4 0.2 0.4 40 10.00000
#> 4 d 9 1 0.1 0.1 90 10.00000
由 reprex package (v0.3.0) 于 2021 年 1 月 2 日创建
我曾经为这种问题打开了一个 feature request,但显然 {dplyr} 的情况太特殊了。当您点击链接时,您还可以找到其他选项来执行此类操作。
,一种选择可能是:
df %>%
mutate(across(c(A,.names = "R_{col}")/across(starts_with("error")))
label A B error_A error_B R_A R_B
1 a 4 10 0.4 0.3 10 33.33333
2 b 3 0 0.3 0.0 10 NaN
3 c 8 4 0.2 0.4 40 10.00000
4 d 9 1 0.1 0.1 90 10.00000
对于这种情况,我发现基本 R 解决方案也很直接且高效。它不需要遍历列或唯一值。您定义两组列并直接将它们分开。
对于您分享的示例,我们可以通过查找仅包含一个字符的列名称来识别 "A" 和 "B" 列。
cols <- grep('^.$',names(df),value = TRUE)
error_cols <- grep('error',value = TRUE)
df[paste0('R_',cols)] <- df[cols]/df[error_cols]
df
# label A B error_A error_B R_A R_B
#1 a 4 10 0.4 0.3 10 33.3
#2 b 3 0 0.3 0.0 10 NaN
#3 c 8 4 0.2 0.4 40 10.0
#4 d 9 1 0.1 0.1 90 10.0

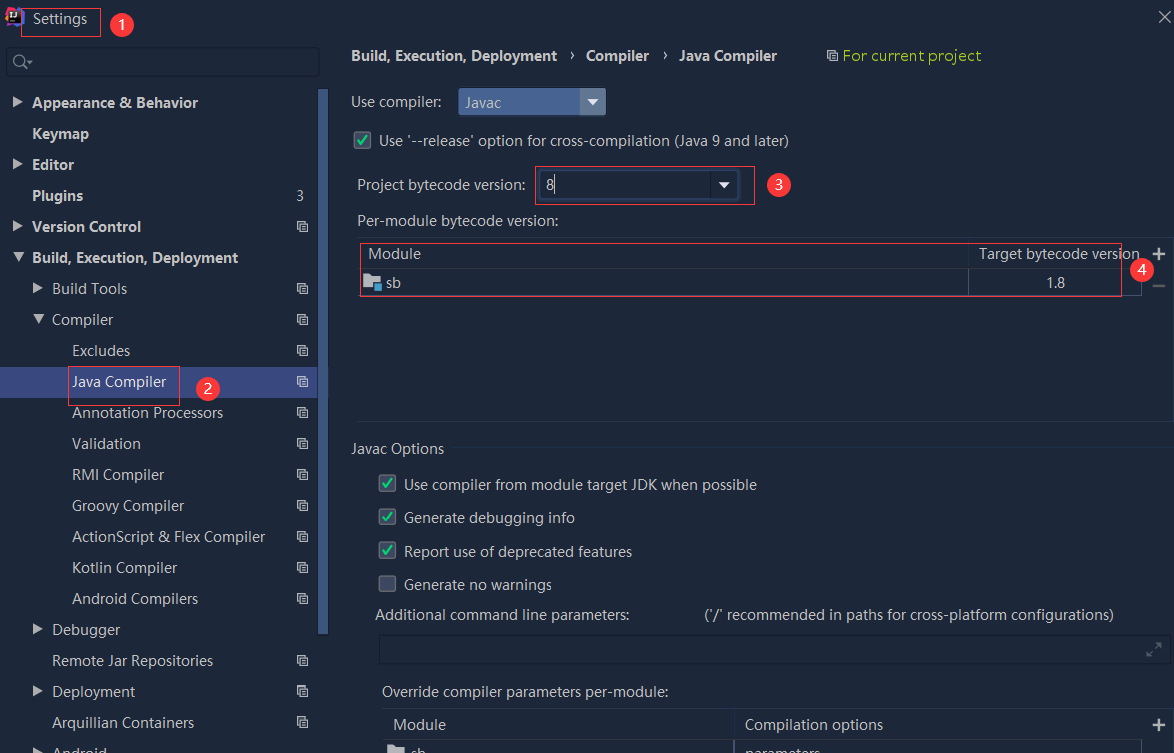 依赖报错 idea导入项目后依赖报错,解决方案:https://blog....
依赖报错 idea导入项目后依赖报错,解决方案:https://blog....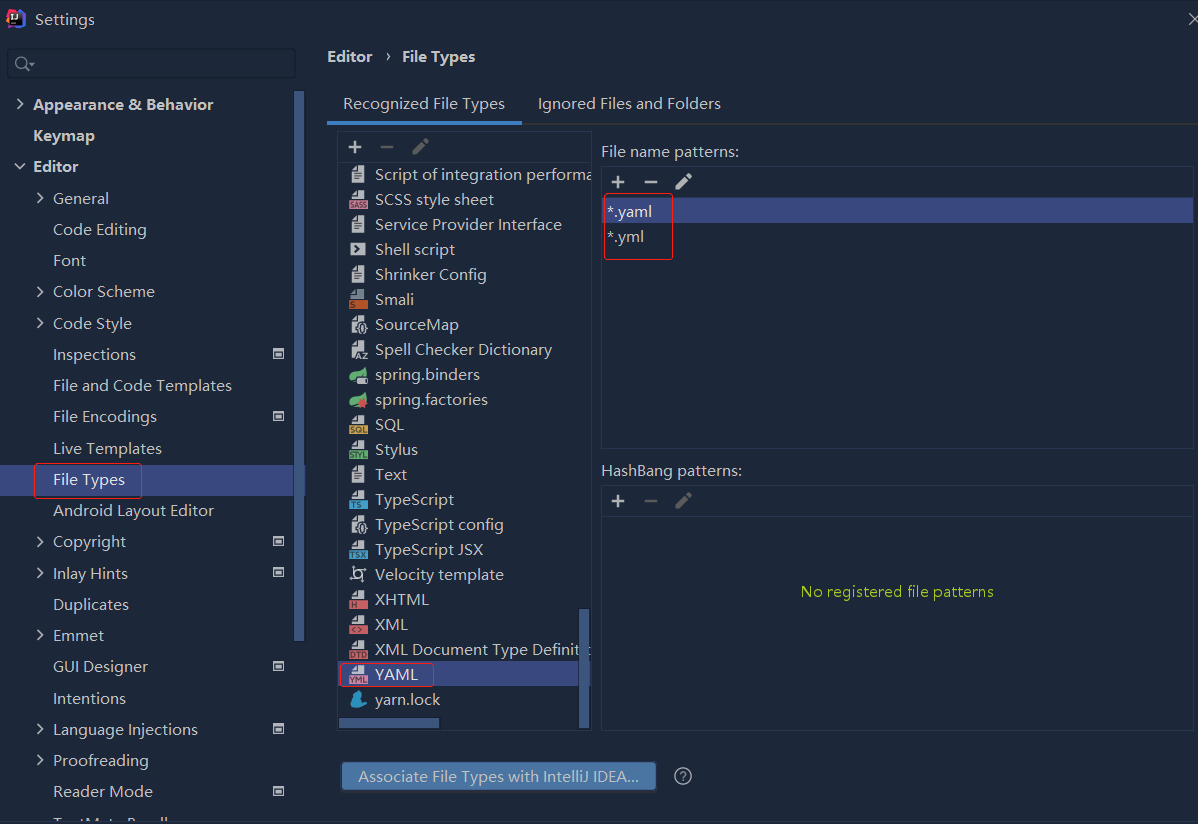
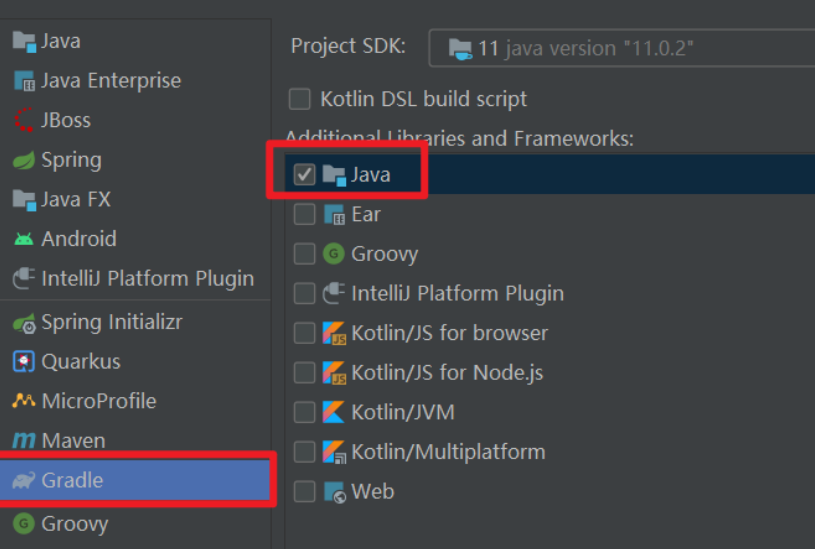 错误1:gradle项目控制台输出为乱码 # 解决方案:https://bl...
错误1:gradle项目控制台输出为乱码 # 解决方案:https://bl...