问题描述
我试图制作一个 perl 代码,以 .fna 文件格式获取 DNA 的 fasta 序列的反向补充。 sequence02C.fna 文件包含数百个 DNA 序列:
>adbca3e
TGCTCCCCACGCTTGCCTCTCCAGTACTCAACCAAAGCAGTCTCTAGAAAAACAGTTTCCAACGCAATACGATGGAATTCCACTTCCCAAATATCTC
>4c2a958
TCCCCACGCTTTCGCGCTTCAGCGTCAGTATCTGTCCAGTGAGCTGACTTCTCCATCGGCATTCCTACACAGTACTCTAGAAAAACAGTTTCTGCTC
>0639b5b
TCGCGCCTCAGTGTCCAACGCAATACGAGTTGCAGACCAGGACACATGGAATTCCACTTCCCTCTCCAGTACTCAACCAAAGCAGTCTCTAGAAAAG
我使用了以下命令,它可以打开文件并反转,但不会在输出中显示序列 ID(例如:>adbca3e)。
代码是:
#!/usr/local/perl
open (NS,"sequence02C.fna");
while (<NS>) {
if ($_ =~ tr/ATGC/TACG/) {print $_;}
}
输出文件只是序列的互补而不是反向。 此外,它不包含序列 ID ">adbca3e"
任何人都可以建议适当的代码来立即执行此序列的反向互补并将结果放入输出文件中吗?
解决方法
您只打印包含 A、T、G 或 C 的行。你想打印每一行,所以打印不应该是有条件的。
#!/usr/local/perl
use strict; # Always
use warnings; # Always
while (<>) {
if (/^>/) { # Only modify lines starting with ">".
tr/ATGC/TACG/;
$_ = reverse($_); # You didn't reverse.
}
print; # Print undconditionally.
}
(tr/// 和 print 默认使用 $_。)
注意我没有打开文件。您可以按如下方式使用该程序:
perl program.pl sequence02C.fna >sequence02C_revcomp.fna
或
perl -i~ program.pl sequence02C.fna
后者就地修改文件。 (小心!先测试一下。不过它确实会备份。)
,你说你有一个程序可以“反转”,但它只是提供补充。也许这对你来说是一个很明显的描述,但对我来说不是很清楚。
如果“反向”是指向后打印字符串,只需使用 reverse 函数。补充我假设正在使用相应的核碱基,这就是您的音译本意tr/ATGC/TACG/。
要修复不打印 ID,只需删除打印语句中的 if 条件。
我要做的就是在这样的小程序中使用菱形运算符:
use strict;
use warnings;
use feature 'say';
while (<>) {
chomp;
unless (/^>/) {
tr/ATGC/TACG/; # transliterate non-ids
my $reverse = reverse; # reverse $_
say $reverse; # do something with $reverse
}
say; # print current line
}
然后你可以像这样使用这个程序:
$ perl program.pl sequence02C.fna > output.txt

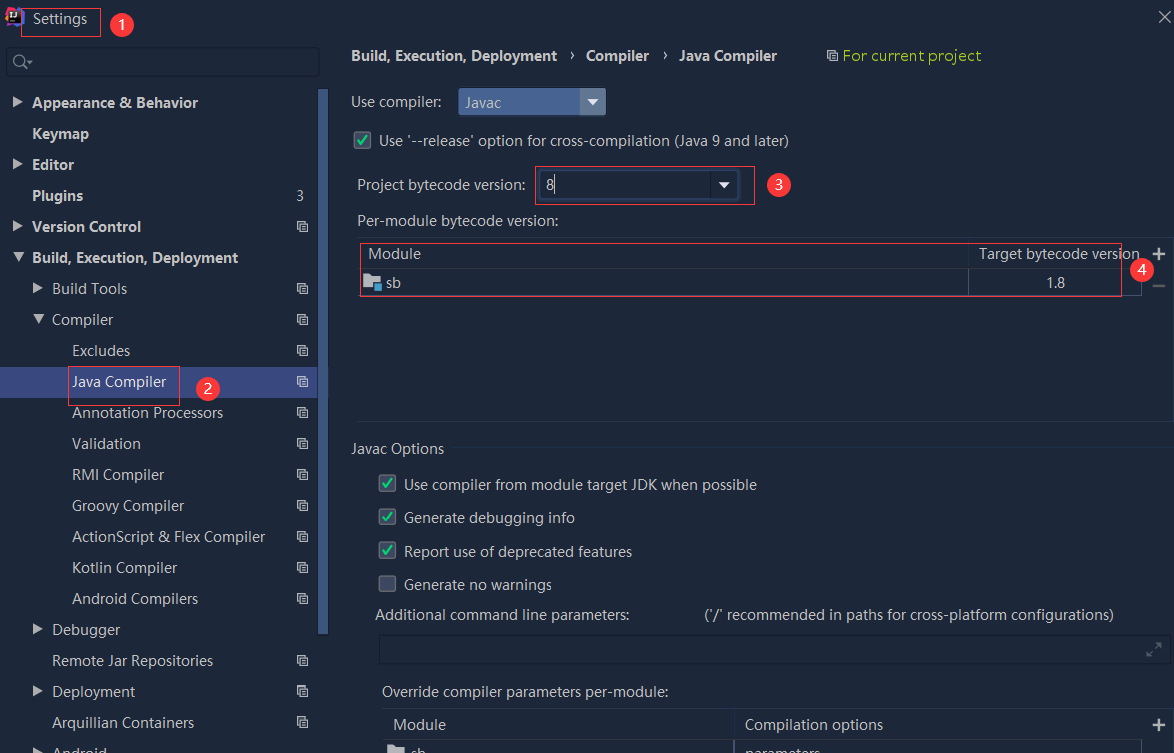 依赖报错 idea导入项目后依赖报错,解决方案:https://blog....
依赖报错 idea导入项目后依赖报错,解决方案:https://blog....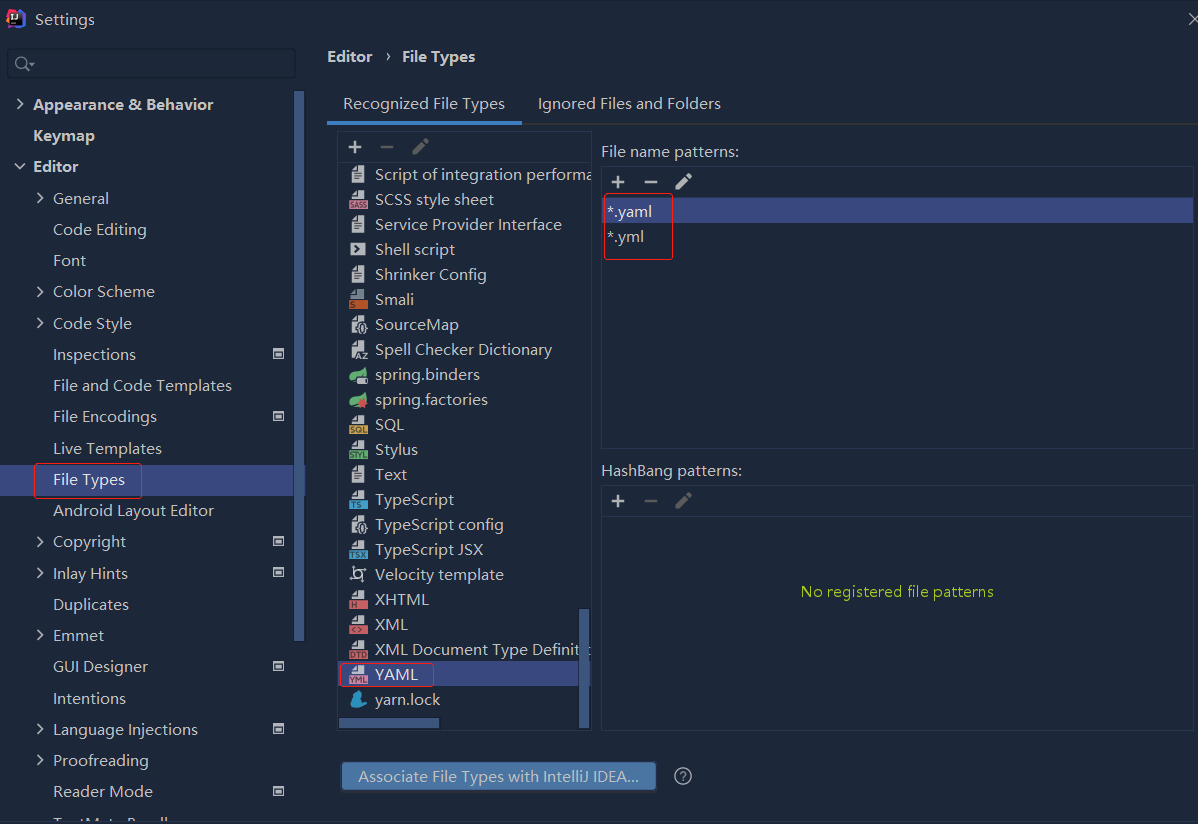
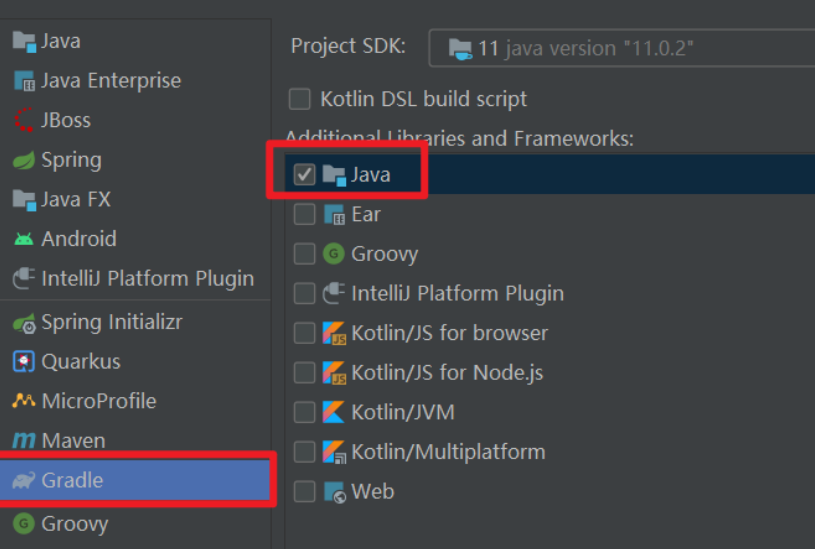 错误1:gradle项目控制台输出为乱码 # 解决方案:https://bl...
错误1:gradle项目控制台输出为乱码 # 解决方案:https://bl...